您当前的位置:检测资讯 > 法规标准
嘉峪检测网 2022-05-25 21:28
摘要:肺炎球菌多糖疫苗或多糖蛋白结合疫苗在预防肺炎球菌性疾病中发挥着重要的作用。肺炎链球菌是肺炎球菌疫苗研制、生产的重要原材料,也是国家重要的战略储备资源,其质量直接或间接影响终产品肺炎球菌疫苗的质量、安全和效力。因此,严格控制肺炎球菌疫苗生产用肺炎链球菌的质量至关重要。本文对肺炎球菌疫苗生产用肺炎链球菌种子批的传统检定方法和分子生物学检定方法(16S rRNA序列测定、基于PCR的血清分型、多位点序列分型、脉冲场凝胶电泳分型和全基因组序列测序)进行概述,并对这些方法的优缺点进行讨论,以期为完善生产用肺炎链球菌种的质控方法和质量标准提供帮助。
关键词:肺炎球菌疫苗;肺炎链球菌;传统检定方法;分子生物学检定方法
Abstract: Pneumococcal polysaccharide vaccine or polysaccharide conjugate vaccine plays an important role in preventing pneumococcal diseases. Streptococcus pneumoniae is an important raw material for the development and production of pneumococcal vaccine, as well as an important national strategic reserve resource. Its quality directly or indirectly affects the quality, safety and efficacy of the final product pneumococcal vaccine. Therefore, it is very important to strictly control the quality of Streptococcus pneumoniae used for the production of pneumococcal vaccine. In this paper, the traditional quality control method and molecular biological method (16S rRNA gene sequencing, PCRbased serotyping, multilocus sequence typing, pulsed field gel electrophoresis and whole genome sequencing) used for the quality control of the seed batch of Streptococcus pneumoniae for pneumococcal vaccine production are summarized, and the advantages and disadvantages of these methods are discussed, in order to provide supports for the improvement of quality control methods and quality standards of Streptococcus pneumoniae for pneumococcal vaccine production.
Key words:pneumococcal vaccine; Streptococcus pneumoniae; traditional quality control method; molecular biological methods
1 引言
肺炎链球菌(Streptococcus pneumoniae)是引起儿童和老年人肺炎球菌性疾病的主要病原菌,其感染可引起急性中耳炎、鼻窦炎、肺炎、脑膜炎、菌血症等[1]。WHO已将肺炎球菌性疾病列为需“极高度优先”使用疫苗预防的疾病。肺炎球菌多糖疫苗或多糖蛋白结合疫苗在预防肺炎球菌性疾病中发挥着重要的作用。国家药品监督管理局网站查询显示,我国有5款23价肺炎球菌多糖疫苗(PPSV23)和3款13价肺炎球菌多糖结合疫苗(PCV13)上市销售。根据肺炎链球菌主要毒力因子荚膜多糖抗原的不同,肺炎链球菌可以分为90多个血清型[2]。其中,最流行的血清型是19F、19A、23F、14、6A和6B型[3]。目前肺炎球菌疫苗生产用菌种包括24个型别,分别是1、2、3、4、5、6A、6B、7F、8、9N、9V、10A、11A、12F、14、15B、17F、18C、19A、19F、20、22F、23F和33F型。肺炎链球菌是肺炎球菌疫苗研制、生产的重要原材料,也是国家重要的战略储备资源,其质量直接或间接影响肺炎球菌疫苗的质量、安全和效力[4]。因此,严格控制肺炎链球菌生产菌种的质量至关重要。
肺炎链球菌生产菌种质量控制应符合《中华人民共和国药典》(简称《中国药典》)2020年版三部“生物制品生产检定用菌毒种管理及质量控制”的有关规定。生产菌种应建立原始种子批、主种子批和工作种子批三级种子批系统。原始种子应验明其历史、来源和生物学特性。工作种子批的生物学特性应与原始种子一致。每批主种子批和工作种子批均应按照国家药品监督管理部门批准的标准和生产规程的要求保管、检定和使用。生产用菌种主种子批检定一般应包括培养特性、革兰氏等染色方法镜检、生化反应、血清学试验、毒力试验、免疫效价测定、培养物纯度、活菌数测定、16S rRNA 序列测定、全基因序列测定等项目[5]。
培养特性、染色镜检、生化反应、胆汁溶菌试验、奥普托欣试验和荚膜肿胀试验等传统的经典肺炎链球菌质量控制方法在菌株的分离、鉴定和质量控制中发挥了重要的作用[6],但传统方法需要活菌操作,对实验人员经验和实验室生物安全级别有较高要求,且存在无法准确分辨菌株之间的差异的局限。近年来,以16S rRNA序列测定、基于PCR的血清分型(PCRbased serotyping)、多位点序列分型(multilocus sequence typing,MLST)、脉冲场凝胶电泳(pulsed field gel electrophoresis,PFGE)分型和全基因组序列测序(whole genome sequencing)为代表的分子生物学方法,以其快速、敏感性高、特异性强以及分辨率高等优点,且能够识别株水平的菌种差异,在细菌鉴定、流行病学调查、系统进化研究等方面发挥了重要的作用,也逐渐应用于肺炎链球菌的分子鉴定中[7,8]。
本文对肺炎球菌疫苗生产用肺炎链球菌种子批的传统检定方法和分子生物学检定方法进行概述,并对这些方法的优缺点进行讨论,以期为完善生产用肺炎链球菌种的质控方法和质量标准提供帮助。
2 肺炎链球菌种子批的传统检定方法
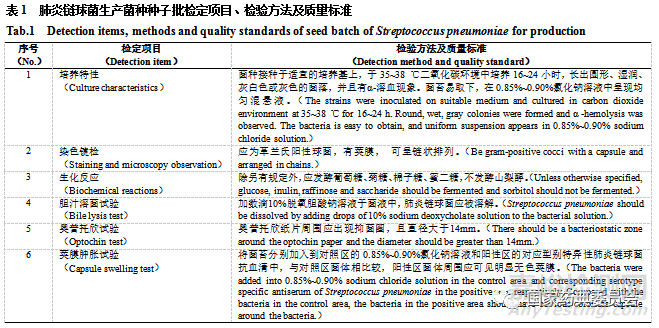
《中国药典》2020年版三部通则中对生物制品生产用菌种的原始种子批、主种子批和工作种子批均有相应的检定要求[5]。表1列出了《中国药典》收录的23价肺炎球菌多糖疫苗种子批的检定项目和相应的检定标准,这些规定对统一和规范疫苗生产企业对肺炎疫苗生产菌种的质量控制,从源头上把控肺炎疫苗的质量起到关键作用。
在实际检验过程中,肺炎链球菌种子批的培养特性、染色镜检、生化反应、胆汁溶菌试验、奥普托欣试验和荚膜肿胀试验,很少出现问题,但这些试验主要依靠检测人员的直接观察,主观性强,对检测人员的经验要求较高。采用糖发酵方法的生化反应鉴定时间持续3~5d。针对生化反应情况,采用梅里埃全自动细菌鉴定试验系统(vitek2)或BIOLOG高通量微生物表型及鉴定分析系统进行生化反应检定,鉴定工作一般可以缩短在24 h内完成。
近年来,国内外均有文献报道鉴定出对奥普托欣(Optochin)耐药的肺炎链球菌[9-12]。Burton等[13]直接使用具有Optochin抗性的肺炎链球菌作为肺炎球菌抗体多重调理吞噬试验(multiplexed opsonization assay, MOPA)的靶菌,用于评价肺炎球菌疫苗的效力。在肺炎链球菌种子批检定中,发现个别企业的某些型别的肺炎链球菌奥普托欣试验抑菌圈直径小于14 mm或者处于临界状态,但胆汁溶菌试验为阳性,表明采用的生产菌种为耐奥普托欣肺炎链球菌。目前,Optochin试验仅作为一种肺炎链球菌鉴定的手段,抑菌圈直径小于14 mm或者处于临界状态,也可能是肺炎链球菌,需要辅助以胆汁溶菌试验进一步证实。肺炎球菌疫苗多为组分疫苗,奥普托欣抗性肺炎链球菌对肺炎链球菌荚膜多糖生产是否具有影响,可否用于肺炎球菌疫苗的生产,还未见相关报道,需要后续进一步验证。
3 肺炎链球菌种子批的分子生物学检定方法
3.1 16S rRNA序列测定
16S rRNA序列测定是通过对细菌16S核糖体RNA(16S ribosomal RNA gene, 16S rRNA )基因特征序列的测定,实现细菌的生物学鉴定。细菌16S rRNA基因全长约1 500 bp,包含9个可变区(variable region,V区)和10个恒定区(Constant region,C区),在结构与功能上具有高度保守性,是细菌分类和鉴定中得到广泛应用的DNA特征序列之一。细菌性疫苗生产用菌种16S rRNA序列测定可参照《中国药典》2020年版四部通则“1021 细菌DNA特征序列鉴定法”进行[14,15]。
《中国药典》方法扩增的是16S rRNA基因V1~V3可变区核酸序列,采用的扩增引物是16SV1F和16SV3R,扩增产物大小约为500 bp。以扩增引物作为测序引物对扩增产物进行序列测定,从而获得扩增产物的核酸序列,核酸序列需去除引物序列,序列方向应与16SV1F引物方向一致。有一些细菌可能在V1~V3可变区外出现基因多态性,对于特定菌种建议使用16S rRNA基因全长引物27F和1492R进行扩增分析[14,16]。16S rRNA序列测定结果判定是“将获得的细菌DNA特征序列与经验证过的专用数据库进行比对,根据比对结果进行判定。”,但未对比对的数据库做出具体规定。目前普遍使用的比对数据库有美国的NCBI数据库(https://www.ncbi.nlm.nih.gov/)和韩国的EzBioCloud 16S数据库(https://www.ezbiocloud.net/)。NCBI数据库收录的16S rRNA基因数据量大,但缺乏有效的验证和筛选。EzBioCloud 16S数据库是经过有效验证和质量筛选的、以收录模式菌株数据为主的数据库。虽然两个数据库比对的结果大多数情况下是一致的,但EzBioCloud 16S数据库可信度更高[17]。
16S rRNA序列测定可以准确鉴定绝大多数菌种的种属,但对同一种属内亲缘关系很近的种分辨率不够。多价肺炎球菌疫苗生产菌种涉及20多个不同血清型别,16S rRNA序列测定可以将生产菌种鉴定到肺炎链球菌,但无法区分各个不同血清型别的疫苗生产菌种[16],如需进一步区分可以进行基于PCR的血清分型。
3.2 基于PCR的血清分型(PCRbased serotyping)
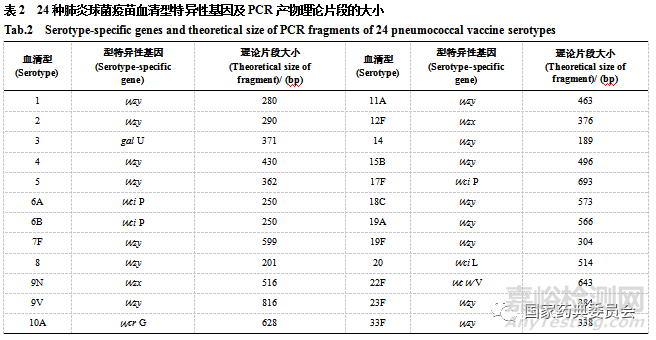
除3型和37型采用合成酶途径(synthase)外,其它所有型别的肺炎链球菌荚膜多糖(capsular polysaccharide, CPS)均是由依赖多糖重复单位聚合酶(Wzy)的途径合成,由位于染色体上的荚膜多糖合成基因簇(cps loci)控制,cps基因簇介于dexB和aliA基因之间[18]。目前已完成90个血清型的cps基因序列测定[19]。cps基因簇的前四个基因(cpsA、cpsB、cpsC和cpsD)在几乎所有血清型中都是保守的,而位点的中心部分包含血清型特异性基因,这些基因是通过基于PCR的方法区分肺炎链球菌的基础。研究人员以cps基因簇为靶基因,设计了一系列引物,建立了基于PCR的检测分析肺炎链球菌血清型的方法。美国疾病预防与控制中心(CDC)官网公布了鉴定41种肺炎链球菌荚膜血清型的引物序列(https://www.cdc.gov/streplab/pneumococcus/resources.html#deductionconventional)。表2列出了24种肺炎球菌疫苗血清型特异性基因及PCR产物理论片段的大小。目前,已经有文献发表基于PCR的血清分型方法适用于肺炎链球菌国家标准菌株的鉴定和质量控制[16]。
与传统的血清凝集血清型分析方法比较,基于PCR的血清型分型方法具有不要求活菌培养、快速、灵敏的特点,但仍存在一些血清型不能精确分型问题,如6A和6B型,20A和20B。由于6A和6B的型特异性基因wci P的大小相同,且6A与6B之间仅是一个单碱基的差别(G584A),PCR血清分型不能准确区分,需要通过wci P基因序列测定进一步区分6A和6B血清型[20-22]。20型肺炎链球菌是肺炎球菌疫苗包含的重要血清型别。研究结果表明,20型肺炎链球菌的荚膜多糖具有六糖重复单位和七糖重复单位2种结构。具有六糖重复单位的20型被命名为20A亚型,七糖重复单位的20型被命名为20B亚型,基于wci L的PCR血清分型虽然能够将20型肺炎链球菌与其他型别分开,但无法区分20A和20B,还需要进行wha F基因测序,对20 型肺炎链球菌进行进一步区分[23]。
此外,由于有些型别的肺炎链球菌血清型特异性基因PCR产物的大小相近,凝胶电泳区分度有限,如果结合分辨率更高毛细管电泳进行PCR产物的分析,能够更加精确的进行血清型的鉴定[24]。
3.3 多位点序列分型(multilocus sequence typing,MLST)
肺炎链球菌MLST分型,即PCR扩增肺炎链球菌7种看家基因(aroE、gdh、gki、recP、spi、xpt和ddl),并将测序得到的7个看家基因序列与肺炎链球菌MLST数据库(https://pubmlst.org/organisms/streptococcuspneumoniae/)进行比对,获得相应的等位基因编号(或者申请新的编号),所有等位基因的编号(aroEgdhgkirecPspixptddl)组成菌株最终的基因序列型(sequence type,ST)。截止到2022年2月2日,MLST数据库已收集了全世界不同国家和地区、不同来源的73 777株肺炎链球菌的MLST信息,共发现283 046种序列型[25]。Zhou M等[26]研究了中国300株侵袭性肺炎链球菌的MLST分布,发现最常见的 ST型是分别是ST320、ST81、ST271、ST876和ST3173。Shi W等[27]从儿童中分离的15群肺炎链球菌中最常见的克隆群(clonal complex)是CC3397、CC6011、CC10088、CC9785和ST8589。Peng S等[28]用MLST方法研究83株肺炎链球菌,发现最常见的ST型是ST271、ST320和ST3397。陈琼等[29-30]通过MLST获得了6群和11群肺炎链球菌疫苗生产用菌种不同的ST型。
MLST分型方法具有准确、快速、易于标准化、便于在不同实验室间进行比较分析的特点,可以检测到菌株特征性的ST,并可反映菌株之间的分子进化关系[31]。不同血清型的肺炎链球菌通常具有不同的ST型,同一血清型内的不同菌株可能具有相同的ST型,所以,当同一血清型的不同生产用菌株具有相同的ST型时,需要采用分辨率更高的方法进行准确鉴定,如脉冲场凝胶电泳(pulsed field gel electrophoresis,PFGE)分型或全基因组测序的方法。
3.4 脉冲场凝胶电泳(pulsed field gel electrophoresis,PFGE)分型
肺炎链球菌脉冲场凝胶电泳是先将肺炎链球菌包埋于琼脂块中,原位裂解胶块中的菌体,再利用限制性核酸内切酶(通常为 Sma I)对菌体全基因组DNA进行酶切,酶切片段进行脉冲场凝胶电泳后,通过专业的聚类分析软件进行电泳图谱和亲缘关系分析的一种分型方法[32-33]。PFGE分型方法因其高分别率,被认为是研究近缘菌株间差异和进化的金标准。研究结果表明相同的血清型或ST型菌株可能具有不同的PFGE图谱[16]。黄洋等[34]用PFGE分型方法获得了24株检测肺炎球菌疫苗功能性抗体的标准株的PFGE图谱,发现各血清群/型的菌株PFGE图谱具有12~15个特征性的条带。PFGE方法准确、重复性好以及分辨率高,非常适合于菌株的流行病学溯源分析,在肺炎链球菌相同血清型和ST型菌株的鉴定上很有优势,其主要不足是操作步骤相对繁琐、实验影响因素多、实验周期较长,不易标准化。
3.5 全基因组序列测序(whole genome sequencing)
近年来,基因组测序技术发展迅速,测序成本逐渐下降,测序周期缩短、测序精度不断提高,二代、三代基因组测序技术逐渐在病原体监测、疫苗等生物制品研发及生产用菌毒种质量控制中得到应用[35-38]。截止2022年2月,NCBI数据库中收录的已完成全基因组测序的肺炎链球菌菌株有9 029株。基因组大小约为2.085 98 Mb,平均GC含量约为39.6%,约编码1951个基因。测序使用的主要方法包括Illumina平台、PacBio平台及最新的单分子Nanopore测序平台[39-41]。采用基因组测序的手段不但可以解析肺炎链球菌本身蕴含的全部遗传信息,还能够全面的了解疫苗应用后肺炎链球菌菌群发生的适应性变化,从而为新型疫苗的研发提供指导。Croucher NJ等[42]利用Illumina HiSeq平台对收集的616株儿童携带的肺炎链球菌进行全基因组测序,发现疫苗免疫接种后,疫苗血清型引起的肺炎球菌疾病的发病率下降,而非疫苗血清引起的疾病增加,出现血清型替换现象。Li L等[43]采用三代Pacific Biosciences 和 Illumina Miseq 测序平台对19A型肺炎链球菌基因组数据进行了比较基因组学分析。Nakano S等[44]对19A型ST320菌株进行了基于基因组数据的系统区域分布研究。Rockett RJ等[45]对采用基因组测序的方法对PCV疫苗应用后19群肺炎链球菌基因组维度的差异进行了分析。Yan Z等[46]用基因组测序的方式对肺炎链球菌血清型和ST型等进行分析,发现最常见的血清型是19F、19A、6B、6A和14,主要的ST型为ST271、ST320和ST90。Gagetti P等[47]采用全基因测序方法研究肺炎链球菌群体结构、ST分型和克隆群(clonal complexes,CCs)。Cuppone AM等[48]使用Nanopore 和Illumina相结合的测序技术测定了肺炎链球菌Rx1的全基因组序列,基因组全长2.03 Mb,有2 054个开放阅读框(open reading frame,ORF), GC含量39.72%。
《中国药典》2020年版三部通则《生物制品生产检定用菌毒种管理及质量控制》中要求建立生产用菌种种子批全基因序列的背景资料,生产用菌种主种子批应进行全基因序列测定。目前,可以利用不同的测序平台在短时间内获得生产用菌毒种全基因组序列结果,但由于测序样本制备的完整性不佳或不同的测序技术所用的基因组拼接算法的不同,可能会导致采用不同测序技术得到基因组序列存在差异,对基因组测序结果的解析造成困难。与病毒相比,建立基于细菌全基因组序列的质量标准比较困难,建议在进行不同批次的菌种全基因组比较时,采用同一种测序技术平台,重点对影响菌种质量或产品质量的关键区域,如荚膜多糖合成位点(cps loci)进行比对分析,针对不同的区域制定相应的质量标准。
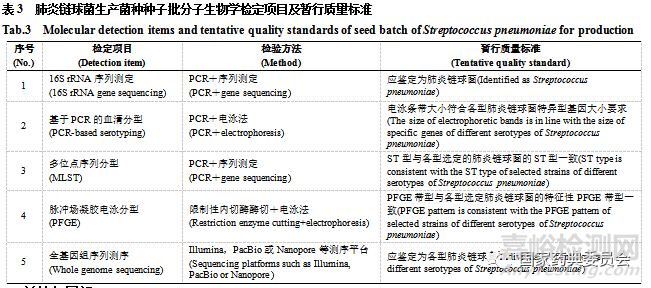
根据各分子生物检测方法的特点,表3列出了肺炎链球菌生产菌种种子批分子生物学检定项目及暂行的质量标准,希望可以作为肺炎链球菌经典的检定方法有力补充,进一步完善肺炎链球菌生产菌种的信息。
4 总结与展望
肺炎球菌多糖疫苗或多糖蛋白结合疫苗在预防肺炎球菌性疾病中发挥着重要的作用。肺炎链球菌是肺炎球菌疫苗研制、生产的重要原材料,也是国家重要的战略储备资源,其质量直接或间接影响终产品肺炎球菌疫苗的质量、安全和效力。因此,严格控制肺炎球菌疫苗生产用肺炎链球菌质量至关重要,应该从菌种的来源、种子批系统的建立及检定、传代次数、生物安全等方面重点关注和全面严格要求。其中,种子批系统的建立及检定又是重中之重。
肺炎链球菌传统的经典的检定方法在各级种子批的检定过程中发挥了重要的作用,对肺炎疫苗的质量起到基础的保障作用[6],但经典的检定方法主要以区分菌株的表型差异为主,需要活菌操作,主观性强,对操作人员的经验有较高要求,不能很好地区分相同表型的不同菌株之间的差别。近年来,逐渐成熟和经过实践验证的分子生物学检测方法,如16S rRNA序列测定、基于PCR的血清分型、MLST分型、PFGE分型及全基因组测序以其快速、敏感性高、特异性强以及分辨率高等优点,逐渐被应用于肺炎链球菌的鉴定和溯源分析中。然而,由于不同分子分型方法的基本原理不同,再加肺炎链球菌的型别众多,可能会导致不同方法对同一菌株的分型结果并不完全一致,且各种方法均存在自身的缺点,例如,16S rRNA序列测定无法区分不同型别的肺炎链球菌;基于PCR的血清型分型方法存在一些血清型不能精确分型问题,如6A和6B型,20A和20B;MLST分型无法区分具有相同ST型的同一血清型的不同菌株;PFGE分型操作步骤相对繁琐、实验影响因素多、实验周期较长,不易标准化;建立基于细菌全基因组序列的质量标准比较困难。因此,在肺炎链球菌生产菌种检定中在传统经典检定方法的基础上,应选用一种或几种分子生物学方法作为生产菌种的补充检定方法,并推进分子生物学方法的标准化,最终形成一套操作简便、分辨力强、灵敏度高、重复性好的生产菌种质量检定方法和标准,确保生产用肺炎链球菌菌种的准确无误,从源头上为肺炎球菌疫苗产品的质量保驾护航。
总之,本文对肺炎球菌疫苗生产用肺炎链球菌种子批的传统检定方法和分子生物学检定方法进行概述,通过对生产用菌种实际检定中出现的相关问题研究探讨,厘清了一些质量控制的薄弱环节,为后续进一步根据不同检验方法的特点制定新的适宜的质量标准提供帮助。
参考文献
[1] 中华预防医学会, 中华预防医学会疫苗与免疫分会. 肺炎球菌性疾病免疫预防专家共识(2020版)[J]. 中华流行病学杂志, 2020, 41(12):1945
Chinese Preventive Medical Association, Vaccine and Immunology Branch of Chinese Preventive Medical Association.Expert consensus on immunoprophylaxis of pneumococcal disease(2020 version)[J]. Chin J Epidemiol, 2020, 41(12):1945
[2] GENO KA, GILBERT GL, SONG JY, et al. Pneumococcal capsules and their types: past, present, and future[J]. Clin Microbiol Rev, 2015, 28(3): 871
[3] WANG Q, SHI W, LI Y, et al. Serotype distribution of Streptococcus pneumoniae isolated from children hospitalized in Beijing children′s hospital (20132019)[J]. Vaccine, 2020, 38(49):7858
[4] 王军志.疫苗的质量控制与评价[M]. 北京:人民卫生出版社, 2013:79
WANG JZ.Vaccine quality control and evaluation[M]. Beijing:People′s Medical Publishing House, 2013:79
[5] 中华人民共和国药典2020年版.三部[S]. 2020:8
ChP 2020. Vol Ⅲ[S]. 2020:8
[6] 陈驰, 肖磊, 石继春, 等. 277株肺炎链球菌生物学鉴定及血清学分型结果分析[J]. 中国医药导刊, 2011, 13(10): 1774
CHEN C, XIAO L, SHI JC, et al. Identification and serotyping result analysis of 277 strains of Streptococcus pneumoniae[J].Chin J Med Guide, 2011, 13(10):1774
[7] 杨越, 叶强.肺炎链球菌分子分型方法研究进展[J]. 中华微生物学和免疫学杂志, 2013, 33(10):789
YANG Y, YE Q.Advances in molecular typing of Streptococcus pneumoniae[J]. Chin J Microbiol Immunol, 2013, 33(10):789
[8] EL AILA NA, EMLER S, KAIJALAINEN T, et al. The development of a 16S rRNA gene based PCR for the identification of Streptococcus pneumoniae and comparison with four other species specific PCR assays[J]. BMC Infect Dis, 2010, 10:104
[9] SOUZA ARV, DE PINA SECM, COSTA NS, et al. Description of optochinresistant Streptococcus pneumoniae due to an uncommon mutation in the atpA gene and comparison with previously identified atpC mutants from Brazil[J]. Sci Rep,2021, 11(1):7936
[10] 王奔, 王晓, 王旭娜, 等. 儿童痰液标本中耐奥普托欣肺炎链球菌的分离及药敏情况[J]. 中国卫生检验杂志,2021, 31(10):1201
WANG B, WANG X, WANG XN, et al. Isolation and drug sensitivity of optopoxinresistant Streptococcus pneumoniae from sputum samples of children[J]. Chin J Health Lab Tec, 2021, 31(10):1201
[11] NUNES S, SÁLEÃO R, DE LENCASTRE H. Optochin resistance among Streptococcus pneumoniae strains colonizing healthy children in Portugal[J]. J Clin Microbiol, 2008, 46(1):321
[12] PIKIS A, CAMPOS JM, RODRIGUEZ WJ, et al. Optochin resistance in Streptococcus pneumoniae: mechanism,significance, and clinical implications[J]. J Infect Dis, 2001, 184(5):582
[13] BURTON RL, NAHM MH. Development and validation of a four fold multiplexed opsonization assay (MOPA4) for pneumococcal antibodies[J]. Clin Vaccine Immunol, 2006, 13(9): 1004
[14] ABELLANSCHNEYDER I, MATCHADO MS, REITMEIER S, et al. Primer, pipelines, parameters: issues in 16S rRNA gene sequencing [J]. mSphere, 2021, 6(1):e0120220
[15] 中华人民共和国药典2020年版.四部 [S]. 2020: 通则1021
ChP 2020. Vol Ⅳ[S]. 2020: General Chapters 1021
[16] 李康, 黄洋, 徐潇, 等.19群肺炎链球菌国家标准菌株分子特征分析及在菌株质量控制中的应用[J].微生物学免疫学进展, 2019, 47(6):20
LI K, HUANG Y, XU X, et al. Molecular characterization of national standard strains of serogroup 19Streptococcus pneumoniae and its application in quality control[J]. Prog Microbiol Immunol, 2019, 47(6):20
[17] YOON SH, HA SM, KWON S, et al. Introducing EzBioCloud: a taxonomically united database of 16S rRNA gene sequences and whole genome assemblies[J]. Int J Syst Evol Microbiol, 2017, 67(5):1613
[18] AANENSEN DM, MAVROIDI A, BENTLEY SD, et al. Predicted functions and linkage specificities of the products of the Streptococcus pneumoniae capsular biosynthetic loci [J]. J Bacteriol, 2007, 189(21): 7856
[19] BENTLEY SD, AANENSEN DM, MAVROIDI A, et al.Genetic analysis of the capsular biosynthetic locus from all 90 pneumococcal serotypes[J]. PLoS Genet. 2006, 2(3):e31
[20] SONG JH, BAEK JY, KO KS. Comparison of capsular genes of Streptococcus pneumoniae serotype 6A, 6B, 6C, and6D isolates[J]. J Clin Microbiol, 2011, 49(5):1758
[21] 陈琼, 李康, 梁丽, 等.应用分子生物学方法鉴定6群肺炎链球菌血清型[J]. 微生物学免疫学进展, 2016, 44(4):12
CHEN Q, LI K, LIANG L, et al. Identification of Streptococcus pneumonia serogroup 6 by molecular biological technology[J]. Prog Microbiol Immunol, 2016, 44(4):12
[22] 陈琼, 陈驰, 石继春, 等.应用PCR 技术和wciP基因测序方法鉴定6群肺炎链球菌血清型[J]. 中华微生物学和免疫学杂志, 2014, 34(4):315
CHEN Q, CHEN C, SHI JC, et al. PCR analysis and wciP gene sequencing for determining the serotypes of Streptococcus pneumonia serogroup 6 strains[J].Chin J Microbiol Immunol, 2014, 34(4):315
[23] 陈琼, 李康, 徐潇, 等.应用whaF基因序列鉴定20型肺炎链球菌血清型[J]. 微生物学免疫学进展, 2016, 44(2):16
CHEN Q, LI K, XU X, et al. Identification of Streptococcus pneumonia serotype 20 with whaF gene sequencing[J]. Prog Microbiol Immunol, 2016, 44(2):16
[24] SELVA L, DEL AMO E, BROTONS P, et al.Rapid and easy identification of capsular serotypes of Streptococcus pneumoniae by use of fragment analysis by automated fluorescence based capillary electrophoresis[J]. J Clin Microbiol, 2012, 50(11):3451
[25] JOLLEY KA, BRAY JE, MAIDEN MCJ. Openaccess bacterial population genomics: BIGSdb software, the PubMLST.org website and their applications[J]. Wellcome Open Res, 2018, 3:124
[26] ZHOU M, WANG Z, ZHANG L, et al.Serotype distribution, antimicrobial susceptibility, multilocus sequencing type and virulence of invasive Streptococcus pneumoniae in China: a sixyear multicenter study[J]. Front Microbiol,2021, 12:798750
[27] SHI W, DU Q, YUAN L, et al.Antibiotic resistance and molecular biological characteristics of non13valentpneumococcal conjugate vaccine serogroup 15 Streptococcus pneumoniae isolated from children in China[J]. Front Microbiol, 2021, 12:778985
[28] PENG S, REN H, DENG J, et al.Genotypic and phenotypic characteristics of Streptococcus pneumoniae from community acquired pneumonia patients and healthy asymptomatic participants in Sichuan province, China[J]. BMC Infect Dis, 2021, 21(1):1030
[29] 陈琼, 龙新星, 李红, 等.6群肺炎链球菌荚膜多糖合成相关基因的分子生物学研究[J].微生物学免疫学进展,2017, 45(4):8
CHEN Q, LONG XX, LI H, et al.Biomolecular analysis of capsular polysaccharide synthesis genes from Streptococcus pneumoniae serogroup 6[J]. Prog Microbiol Immunol, 2017, 45(4):8
[30] 陈琼, 龙新星, 李红, 等.11群肺炎链球菌荚膜多糖合成相关基因的分子生物学研究[J].微生物学免疫学进展,2018, 46(5):49
CHEN Q, LONG XX, LI H, et al.Analysis of capsular polysaccharide synthesis genes from Streptococcus pneumoniae serogroup 11[J]. Prog Microbiol Immunol, 2018, 46(5):49
[31] 钱婧, 薛莲, 谢贵林, 等. 171株儿童侵袭性肺炎链球菌血清分型和多位点序列分型研究[J]. 临床儿科杂志,2012, 30(2): 187
QIAN J, XUE L, XIE GL, et al.Serotyping and multilocus sequence typing of 171 isolates from children with invasive pneumococcal diseases[J]. J Clin Pediatr, 2012, 30(2):187
[32] MCELLISTREM MC, STOUT JE, HARRISON LH. Simplified protocol for pulsed field gel electrophoresis analysis of Streptococcus pneumoniae[J]. J Clin Microbiol, 2000, 38(1):351
[33] 李马超, 张麒, 任红宇, 等.我国部分地区肺炎链球菌血清分型与脉冲场凝胶电泳分型分析[J]. 中国疫苗和免疫,2010, 16(3):265
LI MC, ZHANG Q, REN HY, et al.Analysis on pneumococcus isolated from part of China using serotype and pulsedfield gel electrophoresis[J]. Chin J Vacc Immun, 2010, 16(3):265
[34] 黄洋, 徐潇, 李康, 等.检测肺炎球菌疫苗功能性抗体的标准株分子特征分析[J]. 中华实验和临床病毒学杂志,2021, 35(5):509
HUANG Y, XU X, LI K, et al. Molecular genetic analysis of standard strains for functional antibody detection of pneumococcal polysaccharide vaccine[J]. Chin J Exp Clin Virol, 2021, 35(5):509
[35] BENTLEY SD, LO SW. Global genomic pathogen surveillance to inform vaccine strategies: a decadelong expedition in pneumococcal genomics[J]. Genome Med, 2021, 13(1):84
[36] ALMEIDA SCG, LO SW, HAWKINS PA, et al. Genomic surveillance of invasive Streptococcus pneumoniae isolates in the period prePCV10 and postPCV10 introduction in Brazil[J]. Microb Genom,2021, 7(10):000635
[37] KOZÁKOVÁ J, OKONJI Z, HONSKUS M. Population analysis of Streptococcus pneumoniae serotype 19A by whole genome sequencing in the Czech Republic and in Europe after serotype 19A inclusion in pneumococcal conjugate vaccine[J]. Epidemiol Mikrobiol Imunol, 2021, 70(2):110
[38] NAGARAJ G, GOVINDAN V, GANAIE F, et al. Streptococcus pneumoniae genomic datasets from an Indian population describing prevaccine evolutionary epidemiology using a whole genome sequencing approach[J]. Microb Genom, 2021, 7(9):000645
[39] GOLDEN AR, ADAM HJ, BAXTER M, et al. Whole genome characterization of Streptococcus pneumoniae from respiratory and blood cultures collected from Canadian hospitals before and after PCV13 implementation in Canada: Focus on serotypes 22F and 33F from CANWARD 20072018[J]. Vaccine, 2021, 39(39):5474
[40] SLAGER J, APRIANTO R, VEENING JW. Deep genome annotation of the opportunistic human pathogenStreptococcus pneumoniae D39[J]. Nucleic Acids Res, 2018, 46(19):9971
[41] GARCIAGARCIA S, PEREZARGUELLO A, HENARES D, et al. Rapid identification, capsular typing and molecular characterization of Streptococcus pneumoniae by using whole genome nanopore sequencing[J]. BMC Microbiol, 2020, 20(1):347
[42] CROUCHER NJ, FINKELSTEIN JA, PELTON SI, et al. Population genomics of postvaccine changes in pneumococcal epidemiology[J]. Nat Genet, 2013, 45(6):656
[43] LI L, ZHOU J, LI M, et al. Comparative genomic analysis of Streptococcus pneumoniae strains: penicillin nonsusceptible multidrugresistant serotype 19A isolates[J]. Curr Microbiol, 2022, 79(2):49
[44] NAKANO S, FUJISAWA T, CHANG B, et al. Wholegenome analysisbased phylogeographic investigation of Streptococcus pneumoniae serotype 19A sequence type 320 isolates in Japan[J]. Antimicrob Agents Chemother, 2022, 66(2):e0139521
[45] ROCKETT RJ, OFTADEH S, BACHMANN NL, et al. Genomewide analysis of Streptococcus pneumoniae serogroup 19 in the decade after the introduction of pneumococcal conjugate vaccines in Australia[J]. Sci Rep, 2018, 8(1): 16969
[46] YAN Z, CUI Y, HUANG X, et al. Molecular Characterization based on wholegenome sequencing of Streptococcus pneumoniae in children living in southwest China during 20172019[J]. Front Cell Infect Microbiol,2021, 11:726740
[47] GAGETTI P, LO SW, HAWKINS PA, et al. Population genetic structure, serotype distribution and antibiotic resistance of Streptococcus pneumonia causing invasive disease in children in Argentina[J]. Microb Genom, 2021, 7(9):000636
[48] CUPPONE AM, COLOMBINI L, FOX V, et al. Complete genome sequence of Streptococcus pneumoniae strain Rx1, a Hex mismatch repair deficient standard transformation recipient[J]. Microbiol Resour Announc, 2021, 10(41):e0079921

来源:中国药品标准


